2023年3年3日,我院马为锐实验室与浙江大学医学院徐素宏实验室合作在eLife杂志上发表题为“Enhanced Single RNA Imaging Reveals Dynamic Gene Expression in Live Animals”的研究论文。本研究开发了一种信号放大增强型单分子mRNA成像新技术,并利用该技术揭示了线虫表皮特异基因快速感应细胞膜损伤启动mRNA实时转录的时空动态过程。
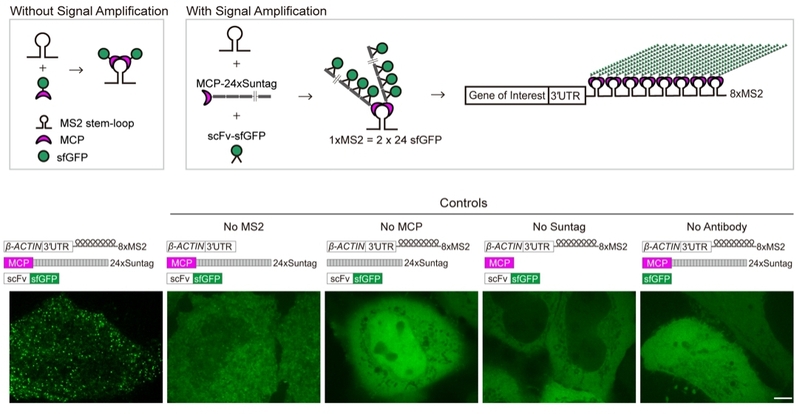
RNA分子在基因表达过程中具有重要的生物学功能且具备高度的动态性。活细胞单分子RNA成像技术使RNA的动态生命历程得以可视化,包括转录、出核、亚细胞定位、翻译以及降解等。MS2-MCP系统被视为活体单分子RNA成像技术中的金标准,并得到广泛使用。MS2系统成像基于MCP蛋白与MS2序列的发夹结构的紧密结合。通过将多个重复的MS2序列插入到目标RNA中,即可招募多个与MCP融合的荧光蛋白,实现单个RNA分子的可视化。MS2系统对内源性mRNA成像需要将24个串联重复的MS2序列(1300 nt)敲入基因组特定位置。由于24xMS2长度长,且为重复序列,定点敲入基因组效率低。为了减少MS2重复数量同时实现更高的RNA成像质量,该研究将MS2与Suntag蛋白质信号放大系统相结合,开发了标记单分子RNA的新技术,命名为MASS(MS2-based signal Amplification with Suntag System)。MASS对单分子mRNA的成像基于插入mRNA 3′UTR中的8xMS2重复序列、融合有串联Suntag的MCP蛋白和识别Suntag的scFV-sfGFP的信号级联放大。该系统实现了高时间分辨率的活细胞及活体动物内源RNA成像。与经典的24xMS2系统相比,该研究使用了8xMS2 (~350 nt),降低了敲入特定基因组位点的难度;同时Suntag招募了更多的荧光蛋白分子,显著放大了单分子RNA的标记信号。MASS技术在细胞和多细胞动物模型中均得到了验证,为单分子RNA亚细胞定位等科学问题的深入研究提供了新方法。
为了检测MASS的可实施性,该研究首先在细胞系中追踪外源b-ACTIN mRNA。3′UTR中插入8xMS2序列的b-ACTIN mRNA结合其他两个成像元件MCP-24xSuntag和scFV-sfGFP,可在细胞中观测到大量显著区别于背景信号的GFP亮点,即b-ACTIN mRNA。缺失任何一个元件(如MS2、或MCP、或Suntag、或scFv),都无法标记mRNA。此外,MASS与单分子荧光原位杂交技术(smFISH)标记信号高度共定位,验证MASS可以准确标记mRNA。尽管先前有研究表明24xMS2影响被标记mRNA的稳定性,但MASS标记并不会产生影响。此外,该研究的多组定量分析结果表明:相比于传统的24xMS2系统,MASS具有更高的信噪比和荧光强度,从而可以对RNA实现长时间且低激光毒性的追踪,为研究单分子RNA动态历程中的机制性问题提供了新的技术手段。
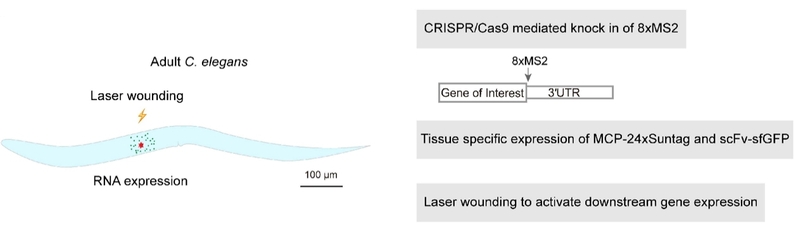
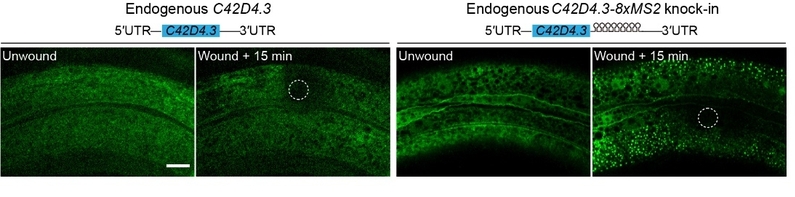
本研究进一步利用线虫模型验证了MASS在活体动物中具有标记mRNA的能力。通过已有的单线虫损伤RNA-seq数据以及qPCR验证,筛选出两个表皮损伤后表达快速上调的基因:C42D4.3和mai-1。本研究构建了C42D4.3与mai-1 的3′UTR区域8xMS2敲入线虫,同时通过组织特异性启动子col-19将MCP-24xSuntag和scFv-sfGFP元件在8xMS2敲入线虫的表皮细胞表达。损伤前野生型(WT)线虫和C42D4.3-8xMS2敲入线虫中几乎没有GFP亮斑;通过激光或针刺损伤线虫表皮,MASS标记的内源C42D4.3和mai-1的mRNA迅速表达,形成大量的点状GFP信号,说明MASS能检测内源mRNA的表达。此外GFP亮斑在损伤后1分钟即出现在损伤区域周围,说明激光损伤能快速激活内源C42D4.3 mRNA表达。随后,GFP亮斑的产生逐渐从损伤区域附近向远端扩展,提示损伤产生的效应因子逐渐扩散并激活基因表达。上述研究表明MASS可用来追踪活体动物中RNA的动态过程以及为动物模型研究RNA转录、迁移等诸多其他科学问题提供了有力的技术支持。

马为锐实验室胡雨岑和浙江大学医学院徐素宏实验室许静秀、高尔罄为本文共同第一作者,马为锐和徐素宏为共同通讯作者。参加该研究的还有马为锐实验室博士研究生范学渊、叶柄成和博士后魏杰丽。该工作受国家重点研发计划、国家自然科学基金委、浙江省自然科学基金、生研院启动基金、杭州市创新团队、浙大青年科研创新专项的资助。
原文链接:https://elifesciences.org/articles/82178

